
細菌不但個體微小,其基因組也比動植物小得多,一般在10M以內。微生物基因組測序能夠檢測微生物全基因組的核苷酸變異,通過全基因組測序,研究人員可以找到大量的單核苷酸多態性位點(SNP)、拷貝數變異(CNV)、插入缺失(InDel)、結構變異(SV)等變異信息,應用范圍涉及臨床醫藥研究、微生物致病性研究、物種進化分析等領域。
1、實驗方案
測序策略:Illumina MiSeq PE250/PE300
數據量:推薦測100×以上
2、數據分析
2.1 標準信息分析
(1)按標準流程進行數據整理及數據質量評估
(2)K-mer分析以及基因組大小估計
(3)序列組裝
(4)組裝基因組的深度和覆蓋度分析
(5)組裝結果的核酸庫比對分析
(6)基因預測
(7)基因功能注釋(GO-COG/KOG)
(8)基因生物通路注釋(KEGG)
2.2 高級信息分析
(1)基因組詳細信息環形圖
(2)共線性分析
(3)基因家族分析
(4)CRISPR預測
(5)基因島預測(毒力島)
(6)前噬菌體預測
(7)分泌蛋白預測
(8)動植物病原菌致病性分析等
3、技術流程

4、案例分析
案例(1)高通量測序分析發現新細菌
德國微生物學與細胞培養研究所微生物系最近從咸水環境微生物墊的低氧過渡區中分離培養獲得了一種中度嗜鹽,專性厭氧,分解糖類的細菌,并編碼代號為L12-Fru-ABT。利用16s測序分析后發現該微生物應該代表著一個新的種屬,研究發現該菌在各種缺/微氧環境中都廣泛存在,比如咸水環境,沸水和動物腸道;隨后,研究人員利用全基因組測序方法對該菌進行基因組序列分析,結合該菌的表型,推斷出L12-Fru-ABT該菌及其相關細菌是能夠專門適應和利用微生物墊或生物膜產生的硫酸化的甘油聚合物。
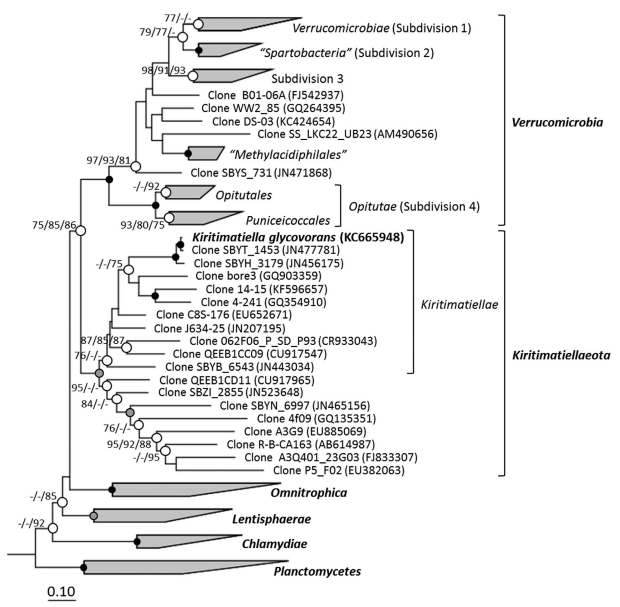
圖1 基于16s rRNA基因測序的L21-Fru-ABT在發育樹的位置圖
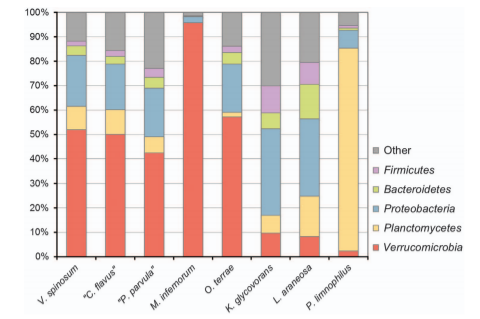
圖2 K.glycovorans L21-Fru-ABT和幾個PVC superphylum的代表性細菌的基因組系統發育構成成分
案例(2)耐鹽耐堿性的克雷伯氏菌.D5A全基因組分析
中國科學院南京土壤研究所研究人員利用Hiseq2000測序平臺,采用Pair-end測序和mate pair測序的技術對克雷伯氏菌.D5A種屬進行了全基因組測序,并對測序數據進行了系統性分析。研究其有利于促進植物生長的相關基因,尤其是與耐鹽耐堿等相關基因。結果發現克雷伯氏菌.D5A種屬的基因組共為5,540,009bp,GC含量占57.15%,同時注釋了一些基因比如3-吲哚乙酸(IAA)合成基因,溶磷作用基因,含體細胞生成基因,羥基丁酮和2,3-丁二醇合成基因,還有固氮功能基因等。
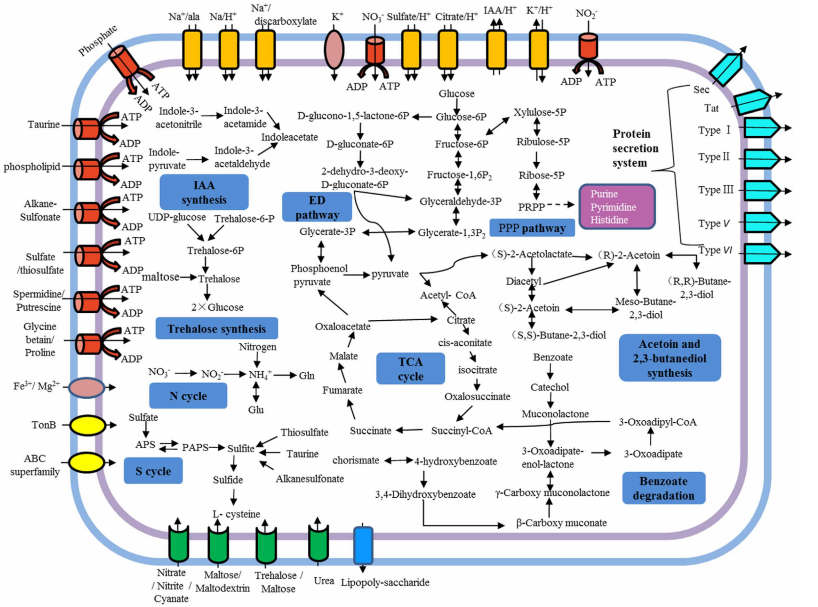
圖1 Klebsiella sp. D5A種屬代謝通路和轉運系統一覽圖
參考文獻
[1] Spring et al. Characterization of the first cultured representative of Verrucomicrobia subdivision 5 indicates the proposal of a novel phylum. The ISME Journal, 2016.
[2] Liu et al. Whole genome analysis of halotolerant and alkalotolerant plant growth-promoting rhizobacterium Klebsiella sp. D5A. Scientific Reports, 2016.